OMEGA PERÚ BLOG |
17 de Marzo 2023
Dispositivos Centrífugos y la Nueva Metodología en el Análisis de Cáncer
Investigadores oncológicos del National Institutes of Health (NIH) desarrollan un novedoso software que facilita los datos de análisis de biomarcadores de vesículas extracelulares.
Las vesículas extracelulares (VE) son pequeñas esferas con membrana que segregan todas las células y que transportan proteínas, ácidos nucleicos, metabolitos, etc. También expresan proteínas en su superficie y pueden utilizarse como biomarcadores traslacionales para ayudar al desarrollo de fármacos y a la investigación terapéutica. La investigación basada en VE es prometedora, pero los estudios se ven obstaculizados por la complejidad de la expresión de marcadores de superficie y por los límites de sensibilidad de los ensayos.
La concentración extremadamente baja de algunas proteínas expresadas dificulta la detección y el análisis diferencial. Para resolver este problema, investigadores del NIH y de otros lugares han unido sus fuerzas para diseñar una herramienta informática de análisis que pueda racionalizar datos complejos y crear un análisis más significativo de las proteínas pertinentes para el cáncer y otras patologías.
Existen kits comerciales para el fenotipado de VE. Los kits de detección de una sola molécula tienen una excelente sensibilidad y pueden evaluar con fiabilidad la presencia de un marcador dado. El problema es que se limitan a informar sobre un color y, por ello, presuponen un conocimiento previo de qué marcadores se están identificando en una muestra.
También existen kits multiplex que pueden detectar varios marcadores al mismo tiempo. Sin embargo, por la enorme cantidad de datos generados puede pasar por alto expresiones de bajo nivel, lo que dificulta la realización de análisis diferenciales.
Los investigadores se propusieron diseñar una metodología que pudiera combinar los puntos fuertes de ambos kits y proporcionar un análisis significativo de un gran volumen de datos con la sensibilidad de una sola molécula. Así, diseñaron MPAPASS (multiplex analysis post-acquisition analysis Software).
El líquido cefalorraquídeo (LCR), el suero y el plasma utilizados en este estudio se obtuvieron de donantes adultos que dieron su consentimiento en el Instituto Nacional de Trastornos Neurológicos y Accidentes Cerebrovasculares. Las muestras de plasma se centrifugaron para eliminar las plaquetas residuales. Para aislar VE de las muestras, estas se incubaron con anticuerpos unidos a microesferas para marcadores comunes de la superficie celular.
A continuación se procedió a la exclusión por tamaño para enriquecer la fracción de VE y a la eliminación de impurezas. Se concentraron 2 mL de muestra de LCR hasta unos 500 mL utilizando un filtro Nanosep® 100kDA de Pall.
Las líneas celulares de cáncer de próstata humano PC3 y PC3pip también se utilizaron como fuente de VEs. Después de cultivar las células, se recogieron alícuotas y se concentraron hasta 5 mL utilizando filtros 100kDA JumbosepTM de 60 mL de volumen. Se recogieron muestras análogas de EV de líneas celulares neurales, renales y de colon humanas normales.
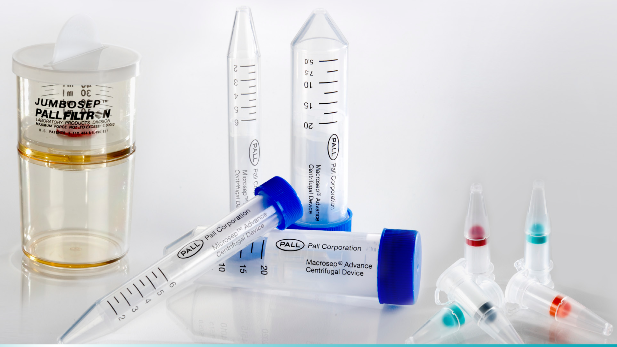
Los dispositivos de filtración centrífuga de Pall están diseñados para evitar la fuga del retentado y la contaminación del filtrado. La filtración es rápida, alcanzando una concentración 50X con una recuperación de muestras superior al 90%. Los filtros centrífugos de Pall están disponibles para volúmenes de muestra que van desde < 50 µL a 60 mL, en una variedad de MWCO (Molecular Weight Cut Off) y composiciones de membrana.
Tras la purificación de las distintas muestras de EV, el equipo de investigación incubó las muestras y luego fueron analizadas mediante citometría de flujo. El software MPAPASS, de nuevo diseño, se utilizó para mejorar la profundidad del conjunto de datos de citometría de flujo.
Uno de los puntos fuertes del método utilizado por los científicos es que el análisis multiplex puede captar un grupo diverso de VE en términos de tamaño y composición. Tanto la intensidad como la variación de la señal proporcionan información útil sobre la abundancia y diversidad de proteínas en una muestra determinada.
Los investigadores utilizaron diversas medidas, incluidos controles de valoración, para normalizar la intensidad de la señal y garantizar la fiabilidad de sus datos. Descubrieron que podían obtener una intensidad de señal lo suficientemente alta de los marcadores de anticuerpos elegidos para poder hacer distinciones entre la intensidad del marcador expresado en muestras EV de dos líneas celulares de cáncer de próstata.
Los científicos fueron capaces de identificar combinaciones únicas de marcadores a partir de diferentes derivaciones de tejido VE y líneas celulares. En conjunto, los resultados del estudio demostraron la utilidad del método de análisis múltiplex para la identificación de muestras y para distinguir métodos de aislamiento. Los investigadores creen que su nuevo software facilitará el intercambio de datos y el tratamiento de muestras de grandes volúmenes de datos.
En el futuro, es razonable esperar que MPAPASS sea muy útil para identificar diferencias clínicas en las muestras, examinar subconjuntos de VE específicos de patologías y agilizar la identificación de nuevos biomarcadores de VE.
Para conocer más sobre los dispositivos de filtrado centrífugo utilizados en este estudio, visite nuestro blog Purificando Ácidos Nucleicos con los Nanosep y la Microcentrifugación
| Conozca más sobre estos equipos en la Categoría “Instrumentacion para ciencias de la vida ” de en nuestra web. Para más información, puede comunicarse con nosotros al 01 336-6523 o ventas@omegaperu.com.pe |
Fuentes:
Pall Corporation. New Methodologies in Cancer Biomarker Analysis. (2022). Pall Laboratory. USA.
Welsh, J. A., Killingsworth, B., Kepley, J., Traynor, T., Cook, S., Savage, J., Marte, J., Lee, M. J., Maeng, H. M., Pleet, M. L., Magana, S., Gorgens, A., Maire, C. L., Lamszus, K., Ricklefs, F. L., Merino, M. J., Linehan, W. M., Greten, T., Cooks, T., Harris, C. C., … Jones, J. C. (2022). MPAPASS software enables stitched multiplex, multidimensional EV repertoire analysis and a standard framework for reporting bead-based assays. Cell reports methods, 2(1), 100136.

