OMEGA PERU BLOG
8 de octubre de 2020
Conoce acerca del uso de dispositivos de ultrafiltración centrífuga Nanosep en el proceso de PCR: antes y después
La capacidad de detectar secuencias específicas de ADN ha sido el núcleo del éxito fenomenal de la biología molecular. Uno de los procedimientos más poderosos y revolucionarios que se utilizan hoy en día para analizar las secuencias de ADN se basa simplemente en la bioquímica única de la replicación del ADN y se denomina Reacción en Cadena de la Polimerasa o PCR.
Sistema de PCR en tiempo real Techne Prime Pro 48; 100-240 V, 50/60 Hz

Esta poderosa técnica permite a los investigadores “amplificar” secuencias de ADN específicas a partir de muestras que contienen solo unas pocas moléculas de ADN individuales con secuencias complementarias a los cebadores (primers). Sin embargo, los métodos de PCR no están exentos de problemas. La extrema sensibilidad de la PCR aumenta la probabilidad de que el cebado falso o la contaminación del ADN conduzcan a la amplificación de la molécula incorrecta.
Una forma de minimizar los artefactos que surgen de las impurezas es trabajar en un entorno extremadamente controlado y purificar los reactivos y los cebadores antes de su uso. Pero incluso en un entorno controlado, la propia muestra puede contener componentes que interfieran con la PCR o provoquen un cebado falso. El uso de dispositivos de ultrafiltración centrífuga Nanosep para eliminar estos contaminantes antes de la PCR puede ayudar a garantizar el correcto resultado.
Dispositivos centrífugos Nanosep® con membrana Omega
Una vez completa una reacción de PCR, el tampón (una mezcla de cebadores y nucleótidos libres) y los fragmentos de ADN recién sintetizados deben separarse para que el ADN sintetizado se pueda utilizar para aplicaciones posteriores. Esta separación se puede lograr por varios medios, como precipitación con alcohol, filtración en gel o ultrafiltración. En este estudio, evaluamos el uso de dispositivos de ultrafiltración centrífuga Nanosep en varios pasos del proceso de PCR: preparación de reactivos y soluciones; purificación / concentración de cebadores; y limpieza y recuperación de productos de PCR.
Análisis experimental
1. Preparación de reactivos y soluciones para PCR
La PCR es un método extremadamente sensible para la detección y amplificación de secuencias de ADN específicas. Esta sensibilidad puede resultar en la amplificación de pequeñas cantidades de ADN contaminante. Este es un problema particular con el ADN antiguo o las secuencias de ADN humano forense porque una molécula contaminante de la piel o célula del cabello de un técnico producirá resultados falsos.
La eliminación del ADN contaminante de los reactivos madre o de las mezclas maestras se puede realizar mediante ultrafiltración. Para asegurarse de que esté libre de ADN, el tampón de síntesis de material final se puede centrifugar en un dispositivo centrífugo Nanosep de 3K o 10K antes de dividirlo en alícuotas en tubos de PCR.
Componentes del Dispositivo Centrifugo Nanosep con Membrana Omega
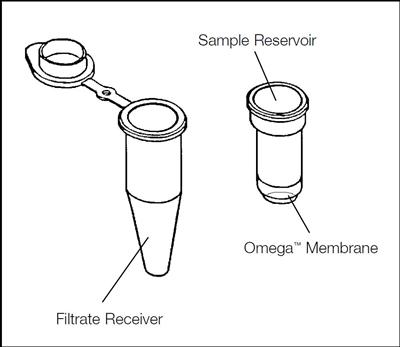
Métodos
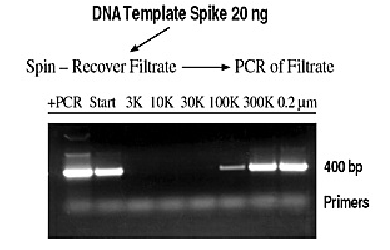
Probamos la capacidad de los dispositivos centrífugos Nanosep para eliminar el ADN contaminante añadiendo 500 ng de plásmido pUC18 a 1,5 ml de TE (Tris 10 mM, EDTA 1 mM, pH 8) antes de dividir la muestra en alícuotas. La muestra se dividió en siete fracciones de 200 µL, una fracción se mantuvo como control (START, 33 ng / uL) y las otras fracciones se centrifugaron en dispositivos centrífugos Nanosep que contenían una selección de membranas de ultrafiltración y microfiltración. Después de la centrifugación (5,000 x g, 10-15 minutos, 25 ° C), se tomó una muestra de 3 µL del filtrado de cada fracción y se agregó a cada reacción de PCR que contenía 45 µL de mezcla de PCR (Invitrogen) que contenía 2 µL de 20 nmoles de pUC18 -mezcla de imprimación complementaria. Se realizó una reacción de PCR de 25 ciclos en condiciones estándar, y se sometieron a electroforesis 25 µL de cada reacción y se tiñeron con bromuro de etidio (Figura 1). Se utilizó una reacción que contenía 200 ng de pUC18 como reacción de PCR control positivo.
Figura 1: Eliminación de la plantilla de ADN de las soluciones madre de PCR
Fuente: Pall Corporation
Resultados y conclusiones
Utilizando una serie de PCR de solo 25 ciclos, se puede hacer una aproximación de la cantidad de molde comparando la intensidad relativa de las bandas de ADN de 400 pb. Como se anticipó, el control positivo tuvo la síntesis más alta. Las muestras experimentales tenían niveles equivalentes de síntesis para el material sin filtrar (START) así como las muestras filtradas a través de los dispositivos de 0.2 µm y 300K, lo que indica que estos dispositivos permitieron que la plantilla introducida pasara completamente. Por el contrario, los dispositivos 3K, 10K y 30K no dieron ningún producto de PCR detectable, lo que indica la eliminación completa de la plantilla de 3.000 pb. El filtrado del dispositivo 100K dio una banda de producto de PCR débil, lo que sugiere que parte de la plantilla pudo pasar a través de este dispositivo.
2. Purificación y concentración de cebadores de PCR
El uso de oligonucleótidos sintéticos ha revolucionado muchos métodos de biología molecular. Son fundamentales para la reacción de PCR porque actúan como cebadores para la síntesis de ADN a partir de puntos de inicio específicos de la secuencia. Los oligonucleótidos se sintetizan utilizando medios químicos. Después de la síntesis, se requieren pasos de purificación para purificar los oligonucleótidos de longitud completa de la mezcla de reacción de síntesis. Antes de su uso en PCR, se requiere desalinización para eliminar los subproductos residuales de los procedimientos de síntesis, escisión y desprotección. La ultrafiltración con concentradores centrífugos es una forma eficaz de desalar y concentrar oligonucleótidos.
Métodos
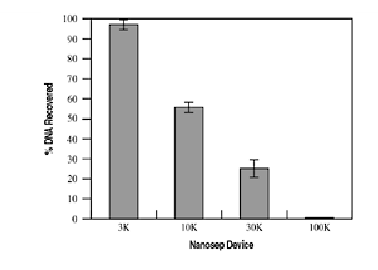
Se evaluó la capacidad de los dispositivos centrífugos Nanosep® para retener los cebadores mediante el filtrado de una solución de 400 µL que contenía 50 ng de oligonucleótidos de 25 pb marcados en el extremo por duplicado utilizando diferentes moléculas dispositivos de corte de peso (MWCO). Estos dispositivos se centrifugaron durante 10 minutos (100 K, 30 K, 10 K) a 30 minutos (3 K) a 5.000 x g. El material retenido se resuspendió en 40 µl de TE, se diluyó a 400 µl y el ADN radiomarcado recuperado se cuantificó usando un contador de centelleo. Se contaron muestras duplicadas de 400 µl del material de partida reunido y se usaron para calcular el porcentaje de recuperación.
Resultados y conclusiones
El dispositivo centrífugo Nanosep 3K retuvo cerca del 90% del oligonucleótido radiomarcado (Figura 2). La elección de un dispositivo de mayor MWCO permitió un tiempo de centrifugado más corto, pero resultó en una disminución de los rendimientos. Si la intención fuera pasar todos los cebadores, entonces el dispositivo de 100K habría sido la mejor opción.
Figura 2: Retención de oligonucleótidos pequeños
Fuente: Pall Corporation
3. Limpieza y recuperación de productos de PCR
La reacción de PCR final puede contener hasta un microgramo de ADN amplificado que se puede utilizar para una variedad de aplicaciones de biología molecular. Estas aplicaciones pueden ser más o menos sensibles al resto de componentes de la mezcla de reacción de PCR. Ciertas enzimas de restricción, así como la ADN ligasa, son particularmente sensibles a la presencia de contaminantes en las muestras de ADN. Debido a que una mezcla de reacción de PCR contiene una variedad de sales, nucleótidos libres, glicerol, proteínas y cebadores, la mayoría de las aplicaciones posteriores requerirán algún tipo de limpieza de PCR.
La separación de los componentes del tampón, los nucleótidos libres y los cebadores del producto de la PCR se puede realizar de diversas formas:
1. Precipitación, utilizando propiedades de solubilidad química para separar selectivamente el ADN. Los principales inconvenientes de utilizar este método para purificar productos de PCR son la pérdida de tiempo y la eliminación incompleta de los componentes del tampón co-precipitante y los contaminantes.
2. Cromatografía, utilizando partículas de exclusión por tamaño o afinidad al vidrio para purificar el ADN de los componentes de la mezcla de PCR. Esta técnica es costosa, generalmente requiere una manipulación significativa y las muestras deben concentrarse después de la elución de la matriz.
3. La ultrafiltración implica el aislamiento y la concentración de productos de PCR utilizando dispositivos de membrana de exclusión por tamaño. Es rápido, requiere muy poca manipulación, los rendimientos son altos, el ADN no se daña y el ADN concentrado está libre de contaminantes que puedan inhibir las reacciones posteriores.
Métodos
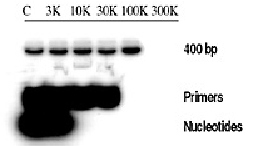
Se realizaron dos experimentos para demostrar el uso de ultrafiltración para la limpieza de reacciones de PCR. El primer experimento mostró la separación de nucleótidos libres, cebadores y productos de PCR. Se añadió una pequeña cantidad de dCTP marcado a la mezcla de reacción de PCR (Invitrogen) y los cebadores se marcaron en los extremos usando dATP. La reacción se ejecutó durante 30 ciclos usando condiciones estándar con una pequeña cantidad de nucleótidos radiactivos y cebadores radiactivos. Se combinaron diez reacciones y se diluyeron alícuotas de 100 µl a 500 µl para centrifugación en una variedad de dispositivos MWCO. El material retenido se recuperó en 20 µL de TE, se sometió a electroforesis usando un gel de poliacrilimida Tris-borato al 10% (BioRad) y se analizó mediante autorradiografía (Figura 3).
Figura 3: Selección de membranas para limpieza de PCR
Fuente: Pall Corporation
El segundo experimento mostró que la ultrafiltración no solo recupera rápidamente los productos de la PCR, sino que los purifica a una calidad superior a la del protocolo de precipitación estándar. Una mezcla combinada de productos de PCR se dividió por la mitad. La mitad se precipitó con 1/10 volumen de acetato de sodio y 2 volúmenes de etanol, se enfrió a -20 ° C durante 2 horas, se centrifugó a alta velocidad durante 30 minutos, se enjuagó con etanol al 70% y se secó al aire antes de la resuspensión en 40 µL de agua. . La segunda fracción se diluyó a 500 µl, se añadió a un dispositivo centrífugo Nanosep de 30K y se centrifugó durante 15 minutos. El material retenido se resuspendió en 40 µl de agua. Ambas muestras de 40 µl se dividieron en fracciones de 10 µl. Una fracción se utilizó como control sin cortar (UC) y las otras se diluyeron en una reacción de digestión de restricción de 20 µl de acuerdo con las instrucciones del fabricante (EcoRI = RI, BamHI = BH1, Xba1 = Xba). Estas muestras se digirieron durante 30 minutos a 37 ° C, se sometieron a electroforesis en un gel de agarosa al 1,5% y se visualizaron las bandas de ADN mediante tinción con bromuro de etidio® (Figura 4).
Figura 4: La desalación por ultrafiltración mejora la actividad enzimática
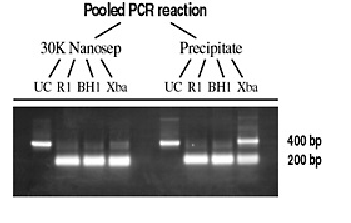
Fuente: Pall Corporation
Resultados y conclusiones
Según los datos de la Figura 3, el dispositivo centrífugo Nanosep de 100K demuestra la mejor combinación de retención de producto de PCR junto con la eliminación completa de cebadores y nucleótidos. Si el deseo es asegurar la eliminación del tampón y los nucleótidos libres, pero no los cebadores, entonces los dispositivos de 30K o 10K retendrán el producto de PCR mientras eliminan los componentes del tampón. Puede ser necesario realizar una limpieza con el dispositivo 30K si el producto de PCR es menor de 200 bp y si la presencia de cebadores no inhibe las aplicaciones posteriores.
Las aplicaciones posteriores, como la digestión de restricción, pueden variar en su sensibilidad a los componentes residuales de la reacción de PCR, dependiendo de la enzima elegida. La enzima de restricción EcoRI tiende a ser resistente y corta el ADN de manera eficaz en condiciones subóptimas, mientras que la actividad de la enzima de restricción Xba1 es extremadamente sensible a la composición del tampón. Los datos (Figura 4) muestran claramente que la enzima Xba1 no puede digerir hasta completar el fragmento de PCR de 400 pb para una muestra que ha sido precipitada. Por el contrario, la muestra que se purificó rápidamente con el dispositivo 30K se digirió completamente usando Xba1.
Este estudio muestra que los dispositivos centrífugos Nanosep se pueden utilizar en aplicaciones de PCR para garantizar productos de PCR de alta calidad. El ADN resultante está presente en forma concentrada y libre de especies moleculares pequeñas que puedan interferir con reacciones posteriores.
Recomendaciones de Equipos y Accesorios:
- Para Cebadores Dispositivo de Centrifugación Nanosep 10K:
- Para la Plantilla de ADN Dispositivo de Centrifugación Nanosep 30K:
- Para la Limpieza del PCR Dispositivo de Centrifugación Nanosep 100K:
- Otros equipos recomendados
Fuente: Pall Corporation. Nanosep® Centrifugal Ultrafiltration Devices and PCR: Before and After
Conoce nuestra sección de dispositivos centrifugos Nanosep con Membrana Omega, de Microcentrifugas y equipos e instrumentos para preparación y análisis por PCR en nuestra web.