OMEGA PERU BLOG
29 de octubre de 2020
¿Sabes cuáles son los 5 métodos espectrofotométricos más conocidos para cuantificación proteica?
Existen numerosos métodos para cuantificar la cantidad de proteína en una muestra, incluidos métodos espectrofotométricos bien establecidos, como los ensayos de Bradford1, Lowry2, Biuret3 y ácido bicinconínico4 (BCA). Además, debido a los aminoácidos aromáticos, las proteínas se pueden medir directamente a 280 nm. Cada uno de estos métodos tiene sus ventajas y desventajas, como se resume en la Tabla 1.
Ensayo
Ventajas
Desventajas
Rango (mg/ml)
BCA
Compatible con detergentes
- Incompatible con agentes reductores.
- Requiere calentamiento
0.2-1.0
Biuret
- Reactivos de fácil preparación
- Compatible con detergentes
- Baja sensibilidad
- Incompatible con sales de amonio
0.5-10
Bradford
- Compatibles con agentes reductores.
- Rápido
- Incompatible con detergente
- Alta variación entre proteínas
0.2-1.4
Lowry
Compatible con detergentes
- Incompatible con agentes reductores
- Reacción de 2 pasos
0.2-1.5
UV
- No requiere estándares.
- No se consume muestra.
Variabilidad entre proteínas
<0.1-3.0
Tabla 1: Resumen de varios métodos espectrofotométricos de ensayo de proteínas
El espectrofotómetro de ciencias de la vida Jenway Genova está preprogramado con parámetros para cada uno de estos ensayos, lo que permite realizarlos de forma rápida y sencilla. Se determinaron las concentraciones de cuatro muestras desconocidas utilizando cinco modos de ensayo de proteínas de Genova.
También demostramos el efecto del detergente y un agente reductor en cada uno de los métodos.
Métodos
Los kits de ensayo de proteínas:
• Reactivo de Bradford – Sigma-Aldrich (B6916).
• Kit de ensayo de proteínas BCA – Sigma-Aldrich (BCA1).
• Kit de ensayo de proteínas Bio-Rad DC – Bio-Rad (500-0111). (Basado en el método Lowry).
Las cuatro muestras desconocidas analizadas fueron las siguientes:
1. Fosfatasa de ácida prostática (PAP) a una concentración nominal de 10 mg/ml (suspensión). Este se diluyó 1 en 10 para todos los ensayos excepto el ensayo de Biuret.
2. Clara de huevo. Esto se diluyó 1 en 100 para todos los ensayos excepto el ensayo de Biuret en el que se diluyó 1 en 10.
3. Albumina de suero bovino (BSA) acetilada, aproximadamente 10 mg/ml. Diluido 1 en 10 para todos los ensayos excepto el ensayo Biuret.
4. Solución estándar de BSA, 1 mg/ml. No diluido. No utilizada en el ensayo Biuret.
Todas las diluciones primarias se realizaron con agua grado biología molecular.
Las muestras se diluyeron luego de la siguiente manera:
a. 1:1 con agua
b. 1:1 con ditiotreitol (DTT) 6 mM
c. 1:1 con 2% TWEEN®20
Esta dilución fue para investigar el efecto del agente reductor DTT y el detergente no iónico TWEEN®20 en cada uno de los diferentes ensayos. Se midieron 100 µl de cada dilución de muestra final con los ensayos BCA, Bradford y Bio-Rad. Para el ensayo de Biuret, se diluyeron 100 µl de la muestra primaria con 150 µl de agua y 250 µl de agua con DTT 6 mM o TWEEN® 20 al 2% para dar un volumen total de muestra de 500 µl.
Para los ensayos de BCA, Bradford y Bio-Rad, se preparó una curva estándar que consta de seis estándares que van desde 0,2 a 1,5 mg/ml de γ-globulina. Para el ensayo de Biuret, la curva estándar osciló entre 0,5 y 3 mg/ml.
Medición
Para cada ensayo colorimétrico, se seleccionó el modo relevante en el Genova y se construyó la curva estándar siguiendo las instrucciones que se muestran en la pantalla. Las concentraciones de la muestra se leyeron directamente siguiendo la generación de la curva estándar. Para el ensayo de Bradford, la longitud de onda se ajustó a 595 nm.
UV directo
Se midieron 100 µl de cada muestra en una microcubeta de cuarzo usando el modo UV directo, usando agua como blanco.
Resultados
Las curvas estándar de cada uno de los ensayos se muestran en la Figura 1.
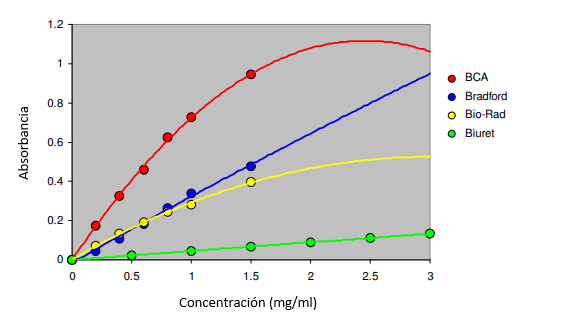
Figura 1: Curvas estándar para cada uno de los ensayos colorimétricos.
Genova utiliza un ajuste de curva polinomial para cada uno de los ensayos; sin embargo, se puede ver que para los ensayos BCA, Bradford y Bio-Rad, las curvas son aproximadamente lineales hasta al menos 1 mg/ml de proteína.
El ensayo de Biuret, aunque menos sensible, es lineal hasta la proteína de 3 mg/ml analizada. La absorbancia varía significativamente entre los ensayos y el ensayo BCA proporciona la señal de absorbancia más alta por encima del fondo, lo que sugiere que es el más sensible de los ensayos colorimétricos probados.
Basándose en las curvas estándar individuales, se determinaron las concentraciones de cada una de las muestras de proteína en cada ensayo. Estos se calcularon de nuevo con los de las muestras sin diluir para que todos los ensayos pudieran compararse directamente. Los resultados se muestran en la Tabla 2.
UV
BCA
Bradford
Bio-Rad
Biuret
PAP
13.98
5.36
7.46
2.18
8.97
Huevo
129.2
131.8
106.0
49.00
123.0
BSA acetilado
7.40
11.20
25.18
3.82
10.90
BSA estándar
0.60
1.07
2.66
0.36
–
Tabla 2: Concentraciones de la muestra original (mg/ml) calculadas a partir de las curvas estándar del ensayo de proteínas y los factores de dilución de la muestra.
El ensayo Bio-Rad proporcionó consistentemente valores más bajos que cualquiera de los otros ensayos. El ensayo de BCA dio valores más cercanos a los esperados para las muestras estándar de BSA y BSA acetiladas. El reactivo de Bradford dio valores aproximadamente dos veces más altos que los esperados para las muestras de BSA.
Para cada muestra se determinaron las proteínas en presencia de DTT 3 mM o TWEEN®20 al 1% para investigar el alcance de la interferencia. Los resultados se muestran en la Figura 2 y demuestran claramente la incompatibilidad de los ensayos de proteína-cobre con el agente reductor DTT, mostrado en rojo. TWEEN®20 también muestra un pequeño efecto en estos ensayos, por lo que siempre es recomendable preparar los estándares en el mismo tampón que las muestras desconocidas. El reactivo de Bradford se ve muy levemente afectado por DTT pero exhibe una absorbancia muy grande en presencia de TWEEN®20. La presencia de DTT y TWEEN®20 también resultó en un aumento de los valores de absorbancia utilizando el método UV directo. Esto es presumiblemente un efecto sobre el plegamiento de proteínas que cambia el coeficiente de absorción de la proteína.
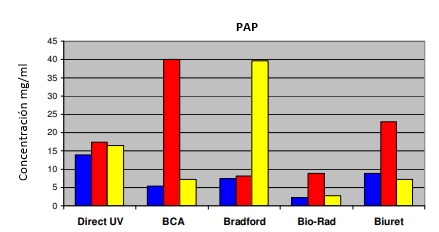
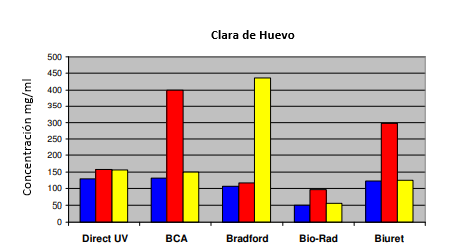
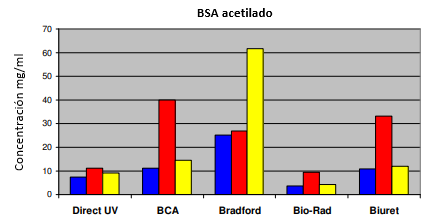
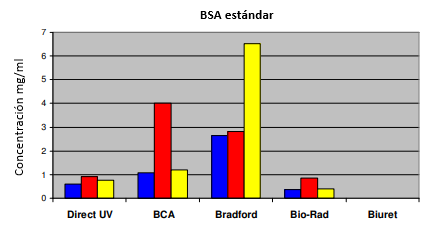
Figura 2: Determinación de proteínas de las cuatro muestras desconocidas sin reactivo añadido (azul), en presencia de DTT 3 mM (rojo) o TWEEN®20 al 1% (amarillo).
Conclusiones
La cuantificación de proteínas a menudo se requiere después de la extracción o como comprobación rápida de fracciones cromatográfcas y antes de análisis posteriores. Jenway Genova está preprogramado para cinco de los métodos más comunes que se utilizan actualmente en la determinación de proteínas. Estos incluyen tanto medidas de absorbancia directa en la región UV como ensayos colorimétricos.
Puntos a considerar son:
2. Presencia de detergentes.
3. Presencia de agentes reductores.
4. Presencia de compuestos quelantes de iones metálicos (EDTA y EGTA).
5. Presencia de sales de amonio.
Siempre es recomendable preparar las diluciones de proteínas estándar en el mismo tampón que la muestra para minimizar los efectos de interferencia.Con los ensayos colorimétricos, cada proteína responderá de forma diferente por su interacción con el tinte o forma de complejos con iones de cobre5.
Por tanto, siempre que sea posible, lo ideal sería que el patrón utilizado fuera una muestra purificada de la proteína que se va a determinar. Como esto no siempre es posible, es importante aceptar que los ensayos representan una estimación más que una cuantificación exacta de la cantidad de proteína presente y que no es posible comparar los datos obtenidos con diferentes ensayos.
Referencias
1. Bradford, M.M. (1976). Anal. Biochem. 72, 248- 254.
2. Lowry, O.H., et al. (1951). J Biol Chem. 193, 265-275
3. Layne, E. Spectrophotometric and Turbidimetric Methods for Measuring Proteins. Methods in Enzymology 10, 447-455, (1957).
4. Smith, P.K., et al. (1985). Anal Biochem. 150, 76-85.
5. http://www.piercenet.com/Proteomics/browse.cf m?fldID=BE21970
Ejemplo de medición con el espectrofotómetro 3 en 1 Jenway Genova Nano
Conozca más sobre nuestros espectrofotómetros y dispositivos centrífugos en la categoría ciencias de la vida de nuestra web. Para más información, puede comunicarse con nosotros al 336 – 6523 o ventas@omegaperu.com.pe


